Hauptmenü
Volunteer Computing
BOINC benutzt vom Anwender nicht genutzte Rechenleistung, um Berechnungen für verschiedene (meist gemeinnützige) Projekte anzustellen und diese dann zurückzugeben.
Mit Hilfe dieser Software kann die Leistung von mehreren Tausenden Rechnern über ein Intranet oder das Internet für ein Projekt zur Verfügung gestellt werden.
Ich bin im Moment in folgenden Projekten eingetragen:
- Asteroids@Home: Ziel des Projekts ist es, Formen und Spin für einen bedeutenden Teil der Asteroidenpopulation abzuleiten. Als Eingabedaten verwenden wir jede verfügbare Asteroidenphotometrie. Das Ergebnis sind konvexe Asteroidenformmodelle mit der Richtung der Spinachse und der Rotationsperiode. (Seit: 02.01.2019)
- ClimatePrediction.net: Projekt der Universität Oxford, mit dem das Klima der nächsten 50 bis 100 Jahre berechnet werden soll, um damit Aussagen über das Ausmaß der globalen Erwärmung machen zu können. (Seit: 16.08.2012)
- Cosmology@Home: Das Ziel von Cosmology@Home ist es ein Modell zu suchen, welches unser Universum am besten beschreibt und eine Auswahl von Modellen zu finden, welche mit den verfügbaren astronomischen partikelphysischen Daten übereinstimmt. (Seit 11.10.2022; im Rahmen der Abschaltung von MLC@Home freiwerdende Ressourcen wurden hierzu umgewidmet).
- DENIS@Home: Projekt San Jorge Universität, mit dem große Mengen elektrophysiologische Herzsimulationen analysiert werden sollen (Aktiv 05/2018 und wieder seit 11.10.2022)
- Folding@Home: Ist ein Projekt der Stanford University, welches die Proteinfaltung und andere Arten von Molekulardynamik simuliert. Der Hauptzweck des Projekts ist die Bestimmung der Mechanismen der Proteinfaltung, d. h. des Prozesses, durch den Proteine ihre endgültige dreidimensionale Struktur erreichen, und die Untersuchung der Ursachen von Proteinfehlfaltungen. Dies ist von Interesse für die medizinische Forschung über Alzheimer, Huntington und viele Formen von Krebs, neben weiteren, anderen Krankheiten. In geringerem Umfang versucht Folding@home auch, die endgültige Struktur eines Proteins vorherzusagen und zu bestimmen, wie andere Moleküle mit ihm interagieren können, was sich auf die Entwicklung von Medikamenten auswirkt.
Hier gab es mal eine BOINC-Version, welche ich im Einsatz hatte - mittlerweile gibt es nur noch eine eigenständige Version. Im Zuge der Covid19-Pandemie habe ich diesen Client auf einer steigenden Anzahl an Nodes in Betrieb genommen. (Seit: 21.04.2020) - MindModeling@Home: Projekt der Wright State University und der University of Dayton (Ohio), welches durch Simulationen eine Vielzahl noch offener Fragen rund um das Gehirn klären soll. (Seit: 18.12.2018)
- Quake-Catcher-Network (QCN): Sucht nach Erdbeben. (Seit 06.07.2009) - aktuell allerdings keine Messsonde vor Ort
- Rosetta@home: Projekt der University of Washington, das dazu dienen soll, Proteinstrukturen und Proteinbindungen aus einer Aminosäuresequenz vorherzusagen. Dabei werden Algorithmen entwickelt und getestet, die eine zuverlässige Strukturvorhersage ermöglichen. Eine akkurate Vorhersage von Proteinstrukturen könnte sich als sehr hilfreich für die Entwicklung von Heilverfahren für beispielsweise AIDS, Krebs, Malaria, Alzheimer und Virenerkrankungen erweisen. Im Rahmen der Covid19-Pandemie habe ich die durch das abgelaufene Seti@Home-Projekt freigewordenen Ressourcen auf Rosetta@home umgezogen.
- SIDock@home: Analysiert Medikamente im Kampf gegen SARS-CoV-2. Gemeinschaftsprojekt von covid.si und dem Karelian Research Center der Russian Academy of Sciences. (Seit: 27.12.2021)
- World Community Grid (WCG): Stellt Rechenkapazität für verschiedene gemeinnützige Projekte zur Verfügung, wird von IBM betrieben. Ich bin an allen Projekten beteiligt. (Seit 06.07.2009)
- Universe@Home: Universe@Home zielt darauf ab, die erste Datenbank des simulierten stellaren Inhalts des Universums zu erstellen, von den frühesten Sternen bis hin zu den exotischsten Schwarzen Löchern. (Seit 07.07.2021)
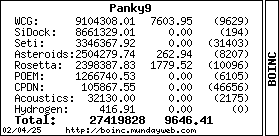
Frühere Projekte
In der Vergangenheit war ich an folgenden Projekten beteiligt (ausgeschieden bin ich meist, weil die Projekte nicht mehr existent sind):
| Name | Beschreibung | Beteiligt ab | Beteiligt bis |
|---|---|---|---|
| Acoustics@home | Beschäftigt sich mit Problemen der Unterwasserakkustik. | 02.01.2019 | ??? |
| APS@Home | APS ist ein Projekt, das sich mit "atmosphärischen Streuungen" befaßt und untersucht, wie sich das auf die Genauigkeit von Klimavorhersagen auswirkt. APS steht für "Atmospheric Process Simulator". |
??? | ??? |
| Hydrogen@Home | Dieses Projekt beschäftigt sich mit sauberen Technologien zur Wasserstofferzeugung mit Hilfe photosynthetischer Bakterien. Hydrogen@Home konzentriert sich dabei auf bisher nicht erforschte Einflüsse von biologischen Katalysatoren auf den Prozess der Wasserstofferzeugung. Ziel ist es eine Effiziente saubere(am besten CO2 freie) Methode der Wasserstofferzeugung zu entwickeln. |
03.07.2010 | ??? |
| MLC@Home | MLC@Home ist ein verteiltes Computing-Projekt, das sich dem Verständnis und der Interpretation komplexer Modelle des maschinellen Lernens widmet, wobei der Schwerpunkt auf neuronalen Netzen liegt. | 07.07.2021 | 02.10.2022 |
| POEM@home | Projekt des Forschungszentrums Karlsruhe, versucht mit verteiltem Rechnen Proteinstrukturen zu optimieren. | 10.07.2009 | 04.10.2016 |
| SETI@Home: | (Auf dem Teleskop "Arecibo" wurde ein zusätzlicher Empfänger angebracht, der das Weltall nach Signalen abscannt, diese werden dann auf den verteilten Rechnern analysiert) (Nehme an den Projekten "SETI@Home Enhanced" und "Astropulse" teil). | 06.07.2009 | 31.03.2020 |
| Virtual Prairie | Agentenbasierte Modellierung eines Ökosystems. | 16.08.2012 | ??? |
Folding@Home
Bei Folding@Home handelt es sich um ein Projekt der Stanford University, welches die Proteinfaltung und andere Arten von Molekulardynamik simuliert. Der Hauptzweck des Projekts ist die Bestimmung der Mechanismen der Proteinfaltung, d. h. des Prozesses, durch den Proteine ihre endgültige dreidimensionale Struktur erreichen, und die Untersuchung der Ursachen von Proteinfehlfaltungen. Dies ist von Interesse für die medizinische Forschung über Alzheimer, Huntington und viele Formen von Krebs, neben weiteren, anderen Krankheiten. In geringerem Umfang versucht Folding@home auch, die endgültige Struktur eines Proteins vorherzusagen und zu bestimmen, wie andere Moleküle mit ihm interagieren können, was sich auf die Entwicklung von Medikamenten auswirkt.
Hier gab es mal eine BOINC-Version, welche ich im Einsatz hatte - mittlerweile gibt es nur noch eine eigenständige Version. Im Zuge der Covid19-Pandemie habe ich diesen Client auf einer steigenden Anzahl an Nodes in Betrieb genommen.
Hier rechne ich im Team Schaffenburg e.V. mit.
 |
 |
 |
 |



